Algorithmus-Detektiv ermittelt passendes Neuro-Modell
In der Forschung kann bereits das Sammeln von Daten schwer sein – doch ein Modell zu finden, welches die Beobachtungen erklärt, stellt oft eine viel größere Herausforderung dar. In der Neurowissenschaft misst man die Zellaktivität von Neuronen. Doch was genau in den zellulären Netzwerken geschieht, kann nur verstanden werden, wenn man die Aktivität durch ein Modell erklären kann. Aber welches Modell passt zu welchen Daten? Eine neuartige Methode, entwickelt von Pedro J. Gonçalves, Jan-Matthis Lueckmann, Michael Deistler, Jakob Macke und Kollegen automatisiert diesen Prozess.
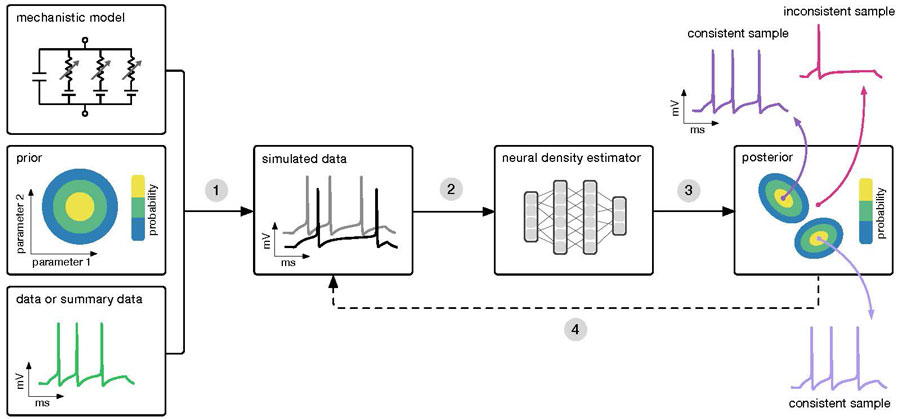
Der Algorithmus (SNPE) benötigt drei Inputs: ein Modell (d.h. Computercode zur Simulation von Daten aus Parametern), Vorwissen oder Einschränkungen der Parameter und empirische Daten. SNPE simuliert eine Reihe von Parameterwerten aus und trainiert ein künstliches neuronales Netz, um jedes Simulationsergebnis auf eine Reihe möglicher Parameter abzubilden. Nach dem Training an simulierten Daten mit bekannten Modellparametern bietet SNPE die gesamte Bandbreite an Parametern, die mit den empirischen Daten und dem Vorwissen übereinstimmen. Grafik von P. Goncalves, J. Lueckmann, M. Deistler and J. Macke
/BN/ caesar, Scherrer et al./
Gehirne bestehen aus einem schier unüberschaubaren Netzwerk aus Nervenzellen. Um die Schaltkreise, die sie formen, zu verstehen, misst man die Aktivität von Neuronen und versucht anhand von Modellen neuronale Mechanismen zu ergründen. Die Identifizierung eines geeigneten Modells, d.h. eines Modells, das mit den gemessenen Daten übereinstimmt, war bisher jedoch oft sehr schwierig, insbesondere bei komplexen, mechanistischen Modellen mit vielen Parametern.
Bisher waren die Möglichkeiten passende Modelle zu finden begrenzt: Entweder wurden Modelle in mühevoller Kleinarbeit so lange modifiziert, bis sie die gemessenen Daten erklären konnten, oder Modelle wurden durch Heuristiken per „brute force“-Prinzip – also dem puren Ausprobieren aller Modelle – bestimmt. Beide Ansätze waren weder systematisch noch effizient.
Pedro J. Gonçalves, Jan-Matthis Lueckmann, Michael Deistler, Jakob Macke und Kollegen fanden eine Lösung für dieses Problem. Sie entwickelten einen Algorithmus, der automatisch anhand der im Experiment gemessenen Daten geeignete Modelle bestimmt. Das Zauberwort dieses Ansatzes heißt „Bayes’sche Inferenz“: Anhand der beobachteten Daten und Simulationen vom Modell identifiziert der Algorithmus alle geeigneten Modelle, die zu den gemessenen Daten passen.
Das wissenschaftliche Arbeiten wird damit enorm beschleunigt. Besonders bemerkenswert an dem neuen Ansatz ist, dass er sehr generell einsetzbar ist – die innere Struktur des Modells spielt keine Rolle. Das ermöglicht es, den Algorithmus auch in anderen Bereichen der biomedizinischen Forschung einzusetzen.
Originalpublikation
Gonçalves, Pedro J, Lueckmann, J., Deistler, M., Nonnenmacher,M., Öcal, K., Bassetto, G., Chintaluri, C., Podlaski, W.F., Haddad, S.A., Vogels,T.P., Greenberg, D.S. and Macke. J.H. “Training deep neural density estimators to identify mechanistic models of neural dynamics”. eLife. Oct. 27. 2020. Doi: 10.7554/eLife.56261




