Maschinelles Lernen verbessert die biologische Bildanalyse
Internationales Forschungsteam entwickelt Algorithmus, der die superauflösende Mikroskopie beschleunigt
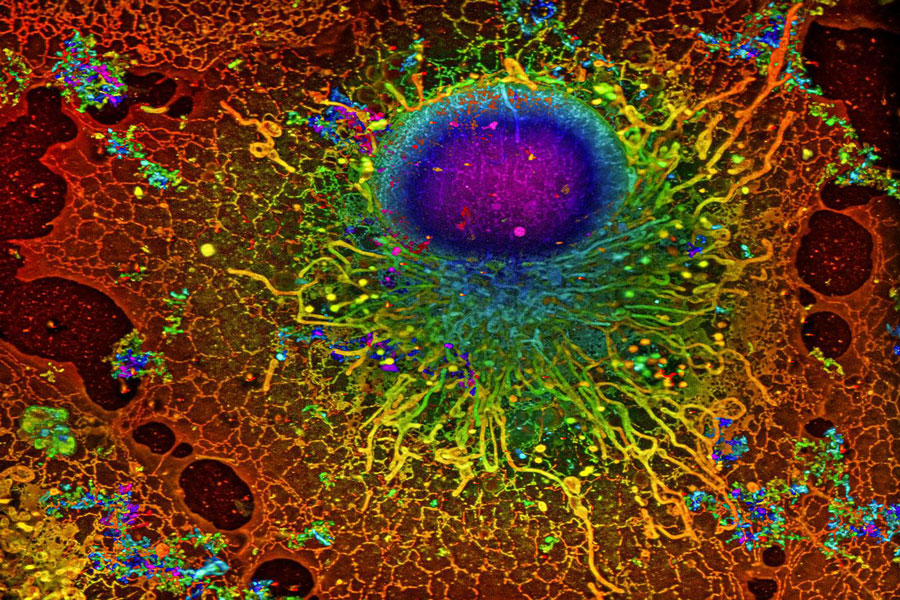
Eine COS-7-Zelle, aufgenommen mit SMLM; Image: Reconstruction by Artur Speiser et. al., data provided by Wesley Legant et. al.
Beteiligtes Bernstein Mitglied: Jakob Macke
/Uni Tübingen/ Mit der superauflösenden Mikroskopie gewinnen Wissenschaftlerinnen und Wissenschaftler neue Einblicke in die Welt der Zellen und können nanometerkleine Strukturen im Zellinneren erkunden. Das Verfahren hat die Lichtmikroskopie revolutioniert und seinen Erfindern 2014 den Nobelpreis für Chemie eingebracht. Tübinger KI-Forscher haben in einem internationalen Projekt einen Algorithmus entwickelt, der diese Technologie wesentlich beschleunigt.
Eine Art der superauflösenden Mikroskopie ist die sogenannte Einzelmolekül-Lokalisationsmikroskopie (single-molecule localisation microscopy, SMLM). Dabei werden die zu erforschenden Strukturen mit fluoreszierenden Molekülen markiert, von denen zu jedem Zeitpunkt nur eine begrenzte Anzahl mit Licht aktiviert wird. Diese lassen sich dann sehr präzise lokalisieren. Mit diesem Trick werden mehrere Bilder einer einzelnen Probe generiert. Diese Rohdaten werden von einem Computerprogramm ausgewertet und zu einem einzigen aussagekräftigen Bild zusammengesetzt. Mit dieser Technik lassen sich viel höhere Auflösungen erreichen als mit der klassischen Lichtmikroskopie. Doch sie hat einen Nachteil: Es braucht eine große Menge an Bildern, was das Verfahren sehr zeitaufwändig macht.
Das Team von Jakob Macke, Professor für Maschinelles Lernen in der Wissenschaft an der Universität Tübingen, hat in einer internationalen Zusammenarbeit einen neuen Algorithmus entwickelt, der diese Einschränkung der SMLM überwindet. Die gemeinsame Arbeit mit der Ries Gruppe des Europäischen Laboratoriums für Molekularbiologie (EMBL) Heidelberg und dem Team von Dr. Srinivas Turaga vom Janelia Research Campus (Virginia, USA) wurde in der Fachzeitschrift Nature Methods veröffentlicht.
Mit Deep Learning zur hochpräzisen Lokalisierung einzelner Moleküle
Der Algorithmus DECODE (DEep COntext DEpendent) basiert auf Deep Learning: Er nutzt ein künstliches neuronales Netz, das durch Trainingsdaten lernt. Statt mit echten Bildern wird das Netz in diesem Fall jedoch mit synthetischen Daten aus Computersimulationen trainiert. Unter Einbeziehung von Informationen über den Aufbau des Mikroskops und der Physik der Bildgebung erzielten die Forschenden Simulationen, die den realen Aufnahmen sehr ähnlich sind. „Das neuronale Netz, das wir mit Simulationsdaten trainiert haben, ist so in der Lage, fluoreszierende Moleküle auch in echten Bildern zu erkennen und zu lokalisieren“, erklärt Artur Speiser, zusammen mit Lucas-Raphael Müller federführender Autor der Arbeit
Einer der Vorteile des Algorithmus DECODE: Er kann Fluoreszenzträger bei höheren Dichten lokalisieren, als bislang möglich, sodass weniger Bilder je Probe benötigt werden. Auf diese Weise kann die Bildgebungsgeschwindigkeit ohne nennenswerten Verlust an Auflösung um das bis zu Zehnfache gesteigert werden. Darüber hinaus kann DECODE die Unsicherheiten quantifizieren ‒ das Netzwerk kann also selbst erkennen, wenn es sich in seiner Lokalisierung nicht sicher ist.
Interdisziplinarität erweitert die Perspektiven der Forschung
„Diese Arbeit ist beispielhaft für den Ansatz unseres Exzellenclusters ‚Maschinelles Lernen: Neue Perspektiven für die Wissenschaft‘“, sagt Macke, dessen Lehrstuhl Teil des Tübinger Clusters ist. „Die Ideen, die diesem Ansatz zugrunde liegen, haben wir ursprünglich in einem ganz anderen Kontext entwickelt. Aber in enger Zusammenarbeit mit Experten für computergestützte Mikroskopie entstanden daraus leistungsstarke Methoden für die Analyse von SMLM-Daten.“ Das Team entwickelte zudem ein Softwarepaket, das den DECODE-Algorithmus verwendet. „Die Software lässt sich einfach installieren und ist kostenlos verfügbar, sodass wir hoffen, dass sie in Zukunft vielen Wissenschaftlern von Nutzen sein wird“, sagt Dr. Jonas Ries vom EMBL.
Originalpublikation
Artur Speiser, Lucas-Raphael Müller, Philipp Hoess, Ulf Matti, Christopher J. Obara, Wesley R. Legant, Anna Kreshuk, Jakob H. Macke, Jonas Ries & Srinivas C. Turaga: Deep learning enables fast and dense single-molecule localization with high accuracy. Nature Methods. https://doi.org/10.1038/s41592-021-01236-x (2021).




